AFV-T: 1000 生物医学機器データ
このチュートリアルでは、生物医学業界の用途を紹介します。腫瘍のある動脈にカテーテルが挿入されます。カテーテルを通じた動脈への薬物注入と、腫瘍への薬物吸収について調べます。
このチュートリアルを実行する前に、<AcuSolve installation directory>\model_files\tutorials\AcuFieldView\AFV_tutorial_inputs.zipから展開したbiomedicalディレクトリを作業ディレクトリにコピーしてください。詳細は、チュートリアルデータを参照してください。
Windowsユーザーの場合、このチュートリアル用に用意されているリスタートを利用するためには、Startメニュー上のAcuFieldViewショートカットのプロパティでStart in欄に値が入力されていないことを確認する必要があります。このプロパティを変更するには、Startメニュー上のAcuFieldViewショートカットを参照し、右クリックしてPropertiesを選択します。Start in欄は、AcuFieldViewのPropertiesダイアログのShortcutタブにあります。この手順が必要な理由は、リスタートファイルで相対パスが使用されることのみであることに注意してください。
図 1.

図 1.
AcuConsoleとAcuSolveを使用した解析
- AcuConsoleを起動します。
- <your working dir>\biomedical\biomedical.acsを開きます。
- AcuSolveを実行して、解を計算します。
- AcuConsoleを終了します。
AcuFieldViewの起動とデータの読み込み
- AcuFieldViewを起動します。
-
View > Background Colorの順にクリックし、白を選択します。
図 2. - Closeをクリックします。
- File > Data Input > AcuSolve [Direct Reader]の順にクリックします。
- Read Grids & Results Dataをクリックします。
- \biomedicalディレクトリを参照し、biomedical.1.logを選択してOpenをクリックします。
- Function Subset Selectionパネル(デフォルトですべての関数が選択された状態で開かれます)で、OKをクリックします。
- データが読み込まれたら、INPUT MODEをAppendに切り替えます。
- biomedical.1.Logを再び読み取って、データがもう一度読み込まれたら、AcuSolve [Direct Reader]パネルを閉じます。
- メインツールバーで、Datasetをクリックします。
- SCALE Xを-1に設定します。
-
Apply、Closeの順にクリックします。
これで、データセット2は、X平面に関してデータセット1を鏡映したものになります。
-
メインツールバーで、Datasetの値を1に変更し、最初に読み込んだデータセットを現在のデータセットとして設定します。
ヒント: Dataset Controlsパネルで現在のデータセットを変更することもできます。
-
Bound
 をクリックして、Boundary Surfaceパネルを開きます。
をクリックして、Boundary Surfaceパネルを開きます。
- Createをクリックし、OSF: Tumor Wallsを選択して、OKをクリックします。
-
DISPLAY TYPEをSmooth shadingに、Geometric COLORINGを赤に変更します。
図 3. - OSF: Artery Wallsで構成される2つ目のサーフェスを、Geometric COLORINGを黄色にして作成します。
-
OSF: Catheter InletおよびOSF: Catheter Wallsで構成される3つ目のサーフェスを、Geometric COLORINGをグレーにして3作成します。
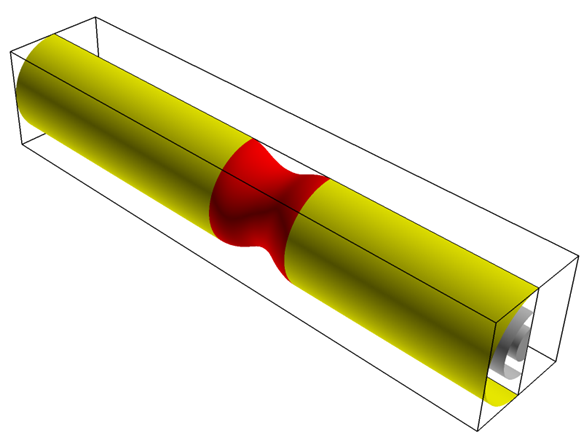
図 4. - File > Save Restart > Current Datasetの順にクリックします。
- \biomedical\restartディレクトリを参照し、このファイルにtumor_1という名前を付けて、Saveをクリックします。
- ツールバーのコントロールを使用するか、DatasetをクリックしてDataset ControlsパネルでIDを変更することで、データセット2を現在のデータセットにします。
-
File > Open Restart > Current Datasetの順にクリックし、tumor_1.datを開いて、データセット1と同じ3つのサーフェスをデータセット2にも作成します。
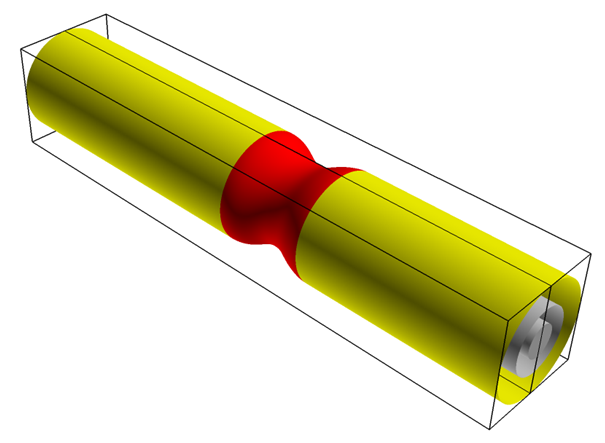
図 5. - 現在のDatasetを1に変更します。
- Boundary Surfaceパネルで、Surface IDを1に設定します。
- 腫瘍壁のVisibilityをオフにします。
- Surface IDを2に設定し、動脈壁のVisibilityをオフにします。
-
Surface IDを3に設定し、Transparencyを50.0%に設定します。
これにより、カテーテルの入口と壁が部分的に透明になります。
-
Transform Controlsツールバーで、
 アイコンをクリックして輪郭表示をオフにします。
アイコンをクリックして輪郭表示をオフにします。
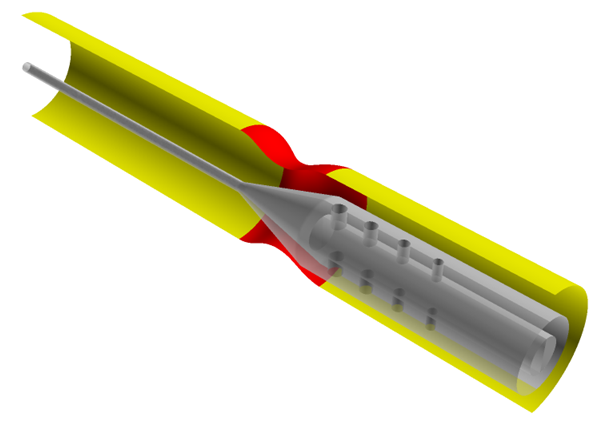
図 6.
流れ場の可視化
この手順では、ベクトル座標サーフェスを作成し、薬物を運んでいる流体と動脈内の血液の相互作用によって生じる流れ場を可視化します。
-
ビューを少し回転して、カテーテルポートと腫瘍にズームします。
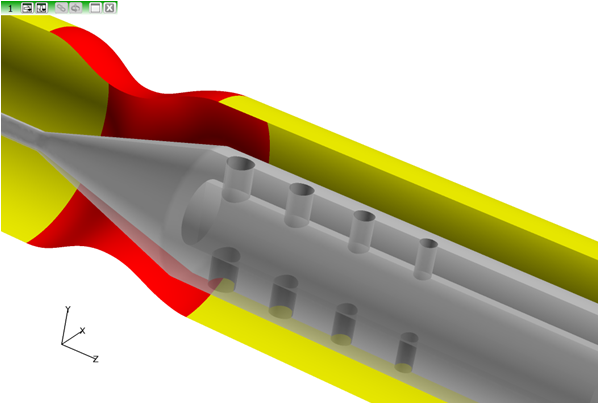
図 7. - File > Open Restart > Formulaの順にクリックします。
- ..\biomedical\restartディレクトリで、bio.frmを選択して、Openをクリックします。
- ツールバーのDatasetをクリックして、Dataset Controlsパネルを開きます。
- Datasetが2に設定されていることを確認します。
-
Coord
 をクリックして、Coordinate Surfaceパネルを開きます。
をクリックして、Coordinate Surfaceパネルを開きます。
- Createをクリックして、COORD PLANEをXに設定します。
- COORD PLANEセクションで、Current positionに-1e-005と入力します。
- DISPLAY TYPEをVectorsに変更します。
- Vector Functionに対して、Selectをクリックします。
- Function Selectionパネルでnrmlz('velocity')を選択し、Calculateをクリックします。
- Coordinate SurfaceパネルでOptionsをクリックし、Vector Optionsパネルを開きます。
- Head Scalingをオンにして、値を0.125に変更します。
- TYPEをTotalからProjectedに変更します。
- Skipオプションをアクティブにして、87.5 %に変更します。
- Length Scaleを0.25に変更します。
- Vector Optionsパネルを閉じます。
-
Geometric COLORINGを白に変更します。
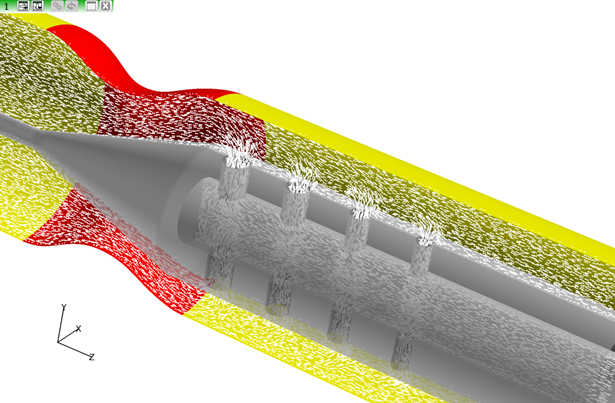
図 8. -
現在のDatasetを1に設定します。データセット1のVisibilityをオフにします。
データセット2の境界サーフェスと座標サーフェスのみが表示されます。
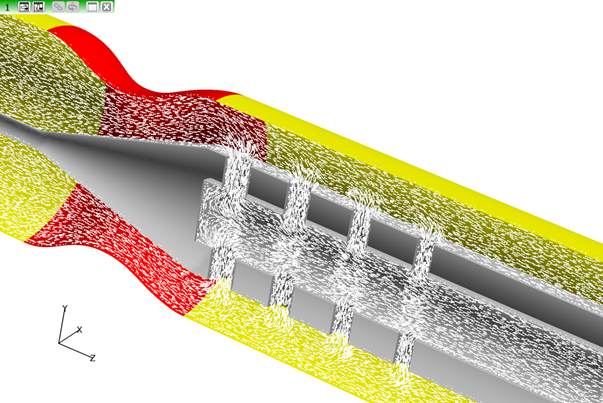
図 9. -
Zoom
 をクリックします。
をクリックします。
-
左マウスボタン(M1)を使用して、いくつかのカテーテルポートを囲むように四角形のズームボックスをドラッグします。ベクトルは、動脈内の血流の方向と速度を示しているとともに、カテーテル内の薬物含有流体の流れを示しています。流体がカテーテルを通って送達ポート内に流れ込む過程で、方向が変化していることに注目してください。薬物含有流体と動脈内血流の間で生じている流れの相互作用にも注目してください。
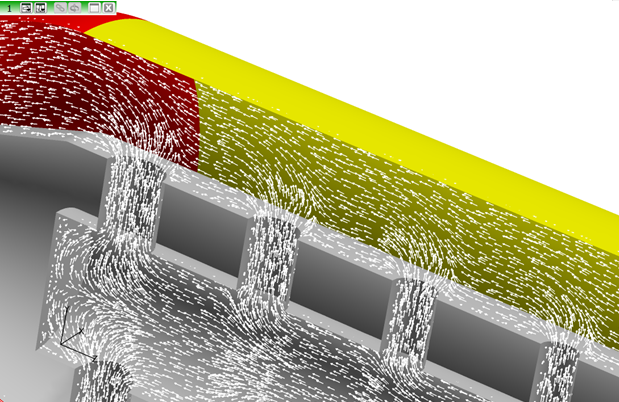
図 10. -
Undo Zoom
 をクリックして、ビューをリセットします。
ヒント: ズームを再び取り消して、以前のズームにビューをリセットできます。ズームを変更するには、マウスの右ボタンを使用して可視化ウィンドウでドラッグします。
をクリックして、ビューをリセットします。
ヒント: ズームを再び取り消して、以前のズームにビューをリセットできます。ズームを変更するには、マウスの右ボタンを使用して可視化ウィンドウでドラッグします。
動脈壁上のせん断の表示
- Artery Wallをダブルクリックしてデータセットを2に設定し、Surface IDが2に設定されたBoundary Surfaceパネルを開きます。
- Scalar Functionに対して、Selectをクリックします。
- Function Selectionパネルで、下にスクロールしてshearを選択します。
- Calculateをクリックします。
- COLORINGをGeometricからScalarに変更します。
- Colormapタブで、最小値と最大値をそれぞれ100と500に変更し、Number of Contoursを32に変更します。
- Filled Contourをオンにします。
-
Tools > Unifyの順にクリックして、現在のデータセット(データセット2)、および同じタイプ(境界)のすべてのサーフェスで、設定した色範囲を使用してせん断が表示されるようにします。
カテーテルの穴を通じた薬剤送達により、動脈壁上でのせん断速度が非常に早くなっていることに注目してください。これは、動脈上で望ましくない大きさのせん断が生じていることを示しています。
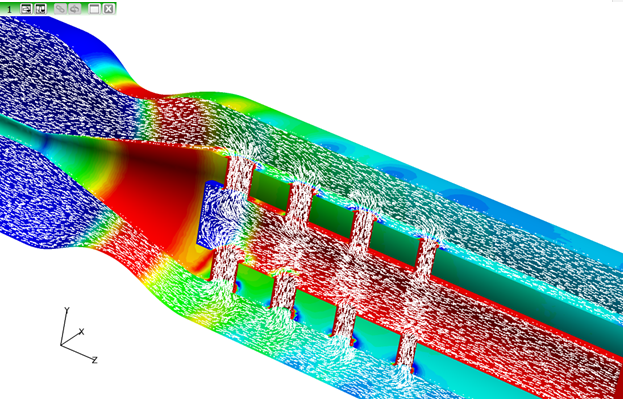
図 11.
応力と集中のコンターの可視化
- ベクトルをダブルクリックして、Coordinate Surfaceパネルを開きます。
- Visibilityをオフにします。
- 動脈のサーフェスをダブルクリックして、Boundary Surfaceパネルを開きます。
- 動脈壁のVisibilityをオフにします。
- Surface IDを1に変更し、腫瘍壁のVisibilityをオフにします。
-
カテーテルの境界サーフェスをダブルクリックして、Geometric COLORINGに変更します。

図 12. - データセット1が現在のデータセットになるようにDatasetを変更して、Visibilityをオンにします。
- データセット1のカテーテルの境界サーフェスをダブルクリックして、Transparencyを0に戻します。
-
データセット1が現在のデータセットになっている状態で、
 をクリックして、Coordinate Surfaceパネルを開きます。
をクリックして、Coordinate Surfaceパネルを開きます。
- 座標サーフェスを作成します。
- Visibilityをオンにします。COORD PLANEをZに、Current positionを-0.0001に設定します。
- Z=-0.0003、-0.0005、-0.0007、および-0.0011に、あと4つの座標サーフェスを作成します。
- 現在のサーフェス(Z=-0.0011)のDISPLAY TYPEをConstantシェーディングに変更します。
- Geometric COLORINGを黒に、ContoursをNoneからScalarに、Scalar Functionをstressにそれぞれ変更します。
- Colormapタブで、最小値と最大値をそれぞれ0.0と180.0に変更し、Number of Contoursを10に変更します。
-
Tools > Unifyの順にクリックして、現在のデータセット(データセット1)、および同じタイプ(座標)のすべてのサーフェスで、設定した色範囲を使用して応力が表示されるようにします。
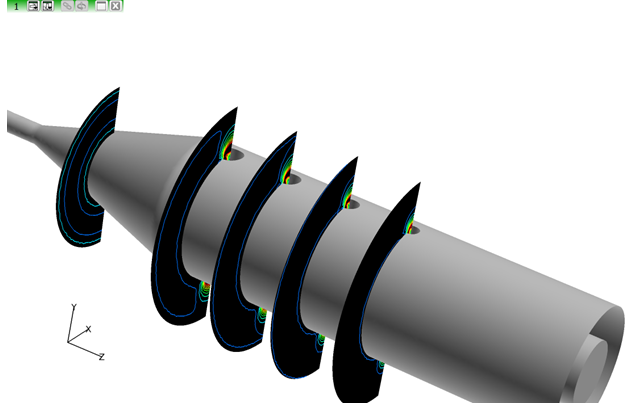
図 13. - File > Save Restart > Current Datasetの順にクリックして、現在のデータセットのリスタートをtumor_2.datという名前で保存します。
- ツールバーでDatasetの値を変更して、データセット2を現在のデータセットにします。
- File > Open Restart > Current Datasetの順にクリックし、現在のデータセットのリスタートを開いて、データセット1と同じ5つのサーフェスをデータセット2に作成します。
- データセット2の1つの座標サーフェスをダブルクリックします。
- Scalar Functionをspecies_1に、COLORINGをScalarに変更します。
-
Tools > Unifyの順にクリックして、この変更を他の4つのサーフェスに伝播させます。
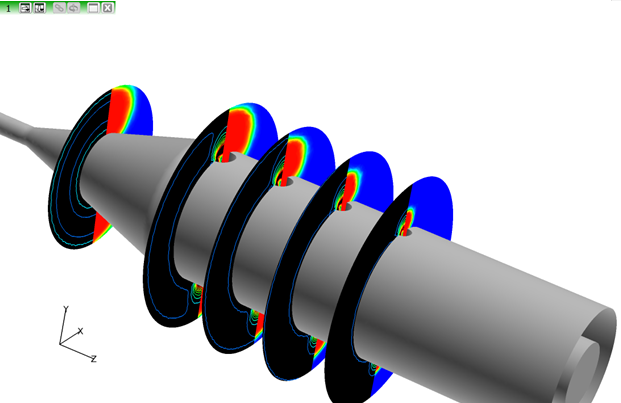
図 14.
質量バランスの計算
- スカラーサーフェスをダブルクリックしてCoordinate Surfaceパネルを開きます。
-
Clear Allをクリックしてから、OKをクリックします。
これにより、一方のデータセット上のすべての座標サーフェスがクリアされます。
- 残りのspecies_1サーフェスのいずれかをダブルクリックして、Coordinate Surfaceパネルを開きます。
-
Clear Allをクリックしてから、Coordinate Surface: Clear All Confirmationパネル上でOKをクリックします。
これにより、他方のデータセット上のすべての座標サーフェスがクリアされます。
- 両方のデータセットの境界サーフェス3について、Visibilityをオンにして、Transparencyを50.0%に変更します。
- データセット2について、境界サーフェス1と2のVisibilityをオンにします。
- データセット2について、4つ目の境界サーフェスを作成します。
- Blood Inletを選択して、OKをクリックします。
- COLORINGをScalarに変更します。
- Scalar FunctionをNconvectiveに変更して、動脈への対流流束を表示します。
- Blood Outletを使用して5つ目の境界サーフェスを作成します。このサーフェスでは、Scalar FunctionがNconvectiveにすでに設定されています。
- ズームアウトしてモデル全体を表示します。
-
ビューを回転して、モデルの上流端を表示します。
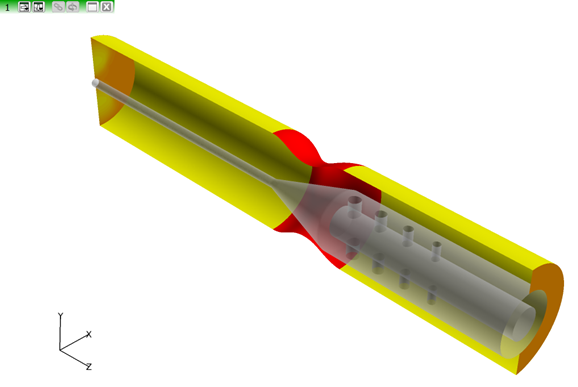
図 15. - Tools > Integrationの順にクリックして、Integration Controlsパネルを開きます。
- Integration ModeをCurrent Surfaceに変更します。
- Integrateをクリックします。動脈Int(S)から出てくる対流流束は約-5.03 e-010です。このサーフェス全体で積分することにより、この動脈から流れ出る薬物の相対量が示されます。

図 16. - 境界サーフェス2(動脈壁)について、COLORINGをScalarに変更し、Scalar FunctionをNdiff-Normalに変更します(壁への拡散流束)。
- Colormapタブで、最小値と最大値をそれぞれ0.0と2000.0に変更します。
-
積分して約2.33e-004という結果を得ます。
このサーフェス上の積分により、動脈壁に衝突している薬物の相対量が示されます。
- 境界サーフェス1(腫瘍壁)に切り替えます。
- COLORINGをScalarに変更し、Scalar FunctionをNdiff-Normalに変更します。Colormapタブで、最小値と最大値をそれぞれ0.0と2000.0に変更します。
- Visibilityをオンにします。
-
積分して約9.77e-005という結果を得ます。
このサーフェス上の積分により、腫瘍壁に衝突している薬物の相対量が示されます。動脈壁と腫瘍壁の積分値の比較により、このモデルでは、動脈壁内に拡散する薬物量は、腫瘍壁内に拡散する薬物量の2倍を上回ることが示されます。
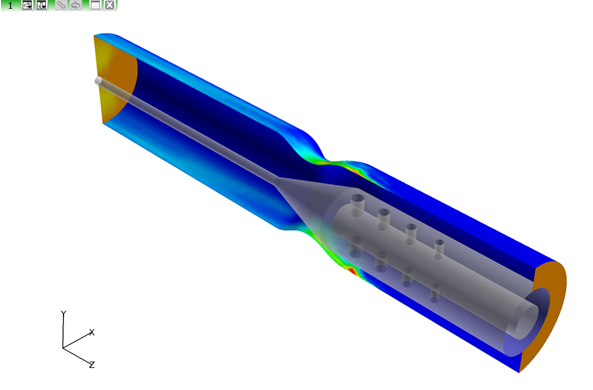
図 17.
薬物送達の可視化
- データセット1の境界サーフェス3(カテーテル入口とカテーテル壁)について、Transparencyを0に設定します。
- 動脈壁(データセット2の境界サーフェス2)をダブルクリックして、COLORINGをGeometric(黄色)に変更します。
-
Tools > Color Mixerの順にクリックするか、ツールバーの
 をクリックします。
をクリックします。
- 黄色いチップをクリックします。Red、Green、およびBlueの値を235、182、および180に変更します。
- Apply、Closeの順にクリックします。
-
データセット1に変更して、Iso
 をクリックし、Iso-Surfaceパネルを開きます。
をクリックし、Iso-Surfaceパネルを開きます。
- Createをクリックします。
- Iso Functionの横のSelectをクリックします。
- Function Selectionパネルでspecies_1を選択し、Calculateをクリックします。
- Iso FunctionのCurrent値を0.5に変更し、色を青にします。
- DISPLAY TYPEをSmoothシェーディングに変更します。
- Color Mixerを開き、青いチップのRGB値を212、212、0に変更します。
-
Apply、Closeの順にクリックします。
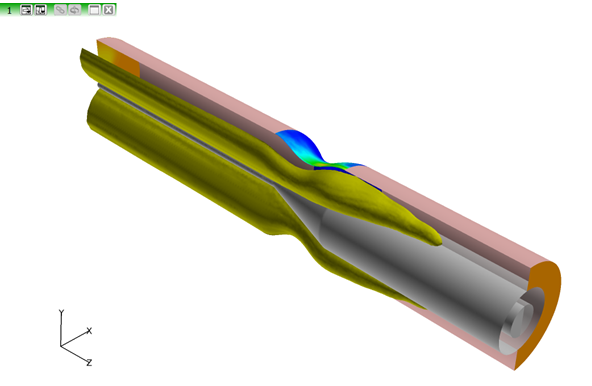
図 18. - 動脈壁と交差しているアイソサーフェスが開かれます。このアイソサーフェスを閉じるには、データセット1で、OSF: Artery Walls、OSF: Tumor Wallsで構成される4つ目の境界サーフェスを作成します。このサーフェスを濃い黄色にします。
- Threshold Functionに対して、Selectをクリックします。
- Function Selectionパネルでspecies _1を選択し、Calculateをクリックします。
- Threshold Clipをオンにして、Minを0.5に設定して“上部の開口部”を埋め、残り部分を切り捨てます。
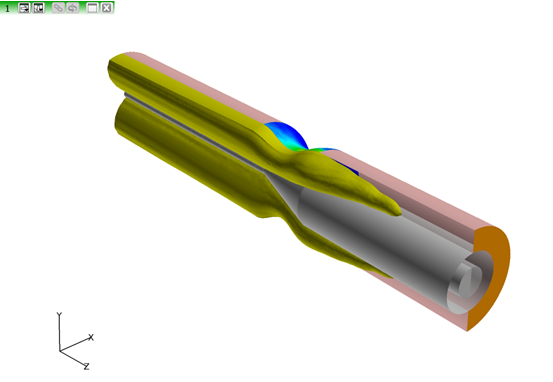
図 19.